Progenesis QI 蛋白质组学与蛋白定量和蛋白鉴定软件为相同的 MSe 数据提供不同的 ID - WKB29713
故障描述
- 在 PLGS3.0.3 和 Progenesis QI Proteomics v3 或 v4 中使用相同的处理参数处理相同的数据时,获得的蛋白质鉴定结果不同
- 确认所有处理参数(例如,Apex3D 低能量和高能量阈值、锁定质量数)相同
- 所有搜索参数都相同(相同的 fasta 文件、酶切试剂、肽和碎片离子质量数容差、允许的修饰、允许的漏切位点数)
环境
- PLGS3.0.3
- Progenesis QI for Proteomics v3 或 v4
注:如果比较 PLGS3.0.3 和 Progenesis QI for Proteomics v4,请确保文章 13412 中描述的 Peptide3D 修饰已应用于 PLGS3.0.3。缺省情况下,此修改在 Progenesis QI for Proteomics v4 中实现,并且可能会对已鉴定的蛋白质产生影响。
原因
处理的保留时间窗口内的差异
在 Progenesis QIp 中设置 MSe 实验时,有两个地方必须指定保留时间范围。对于 MSe ID 搜索,必须在 MSe 数据导入向导中定义保留时间窗口。对于 Progenesis 差分引擎,用户可以在 Automatic Processing(自动处理)向导中定义保留时间窗口。如果只定义第二个,则在 Automatic Processing(自动处理)向导中定义的保留时间窗口不会应用于识别蛋白质的 MSe 峰处理。这意味着所有数据都由 Apex3D 和 Peptide3D 处理并用于 iadbs 搜索,因此 Progenesis QIp 中的搜索未使用 PLGS 中使用的相同数据子集。但是,Automatic Processing(自动处理)中的保留时间限制会正确应用于定量的数据。
解决方法
使用 PLGS 处理参数中使用的相同 RT 窗口重新处理 Progenesis QIp 中的数据。
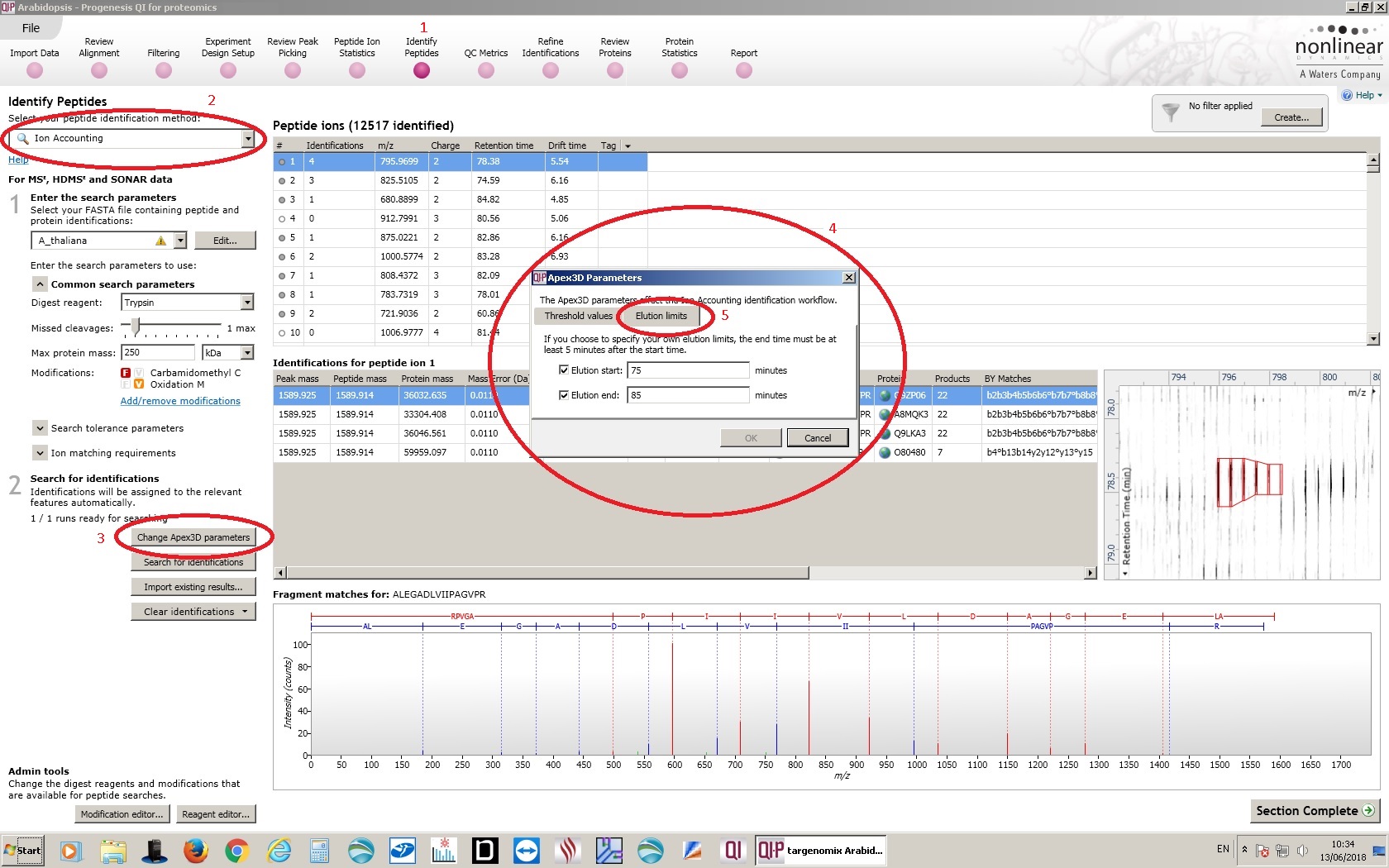
- 导航至 Progenesis QI for Proteomics 中的 Identify proteins(鉴定蛋白质)选项卡。
- 确保 Ion Accounting(离子计数)为选定的肽鉴定方法。
- 单击 Change Apex3D Parameters(更改 Apex3D 参数)按钮。
- 此操作将打开 Apex3D 参数向导,该向导大致相当于 PLGS 中的 Processing(处理)参数。确保 Threshold(阈值)与 PLGS 上使用的值相同。
- 选择 Elution limits(洗脱限制)选项卡,设置与 PLGS 中处理参数相同的保留时间窗口。
- 单击 OK(确定)重新处理数据。注:重新处理数据时,您将看不到进度条。您可以在 Windows“任务管理器”中监视 Apex3D 和 Peptide3D 的进程。这可能需要很长时间。
- 分析中所有样品的 Apex3D 和 Peptide3D 都已完成后,单击 Search for Identifications(搜索标识)按钮,对定义保留时间窗口中的数据重复搜索步骤(相当于 PLGS 工作流程)。
附加信息
为了确保进行同类比较,必须使用正确版本的 PLGS 和 Progenesis QI for Proteomics;否则,使用的处理可执行文件版本将不同。在处理可执行文件方面:
Progenesis QIp v4 = 带有 WKB13412 中所述的 Peptide3D 修饰的 PLGS3.0.3
Progenesis QIp v3 = 不带上述修饰的 PLGS3.0.3
“回滚”MSe 处理程序的 Progenesis QIp v3 = PLGS3.0.2
Progenesis QIp v2 = PLGS 3.0.2
id29713, lockmass, retention time, SUPPLGS