自定义氨基酸修饰无法用于向前查找或向后查找设置指定的残基 - WKB118646
文章编号: 118646点击此处访问英文版本文章
故障描述
- 尽管通过向前查找和向后查找序列应用了自定义氨基酸修饰,确保在特定的蛋白质序列中仅修饰特定残基,但搜索结果中的其他氨基酸也可能会被分配修饰。
- 例如,组蛋白 H3 的“尾部”包含八个赖氨酸 (K) 残基,每个残基都可能被乙酰化。不同的残基修饰会具有不同的生物学效应,这些赖氨酸也可能受到其他修饰的影响(请参阅 The Role of Histone Tails in the Nucleosome: A Computational Study, Erler et al., Biophysical Journal, 107, Vol 12, 2014)。
- 如果想要定义特定于 K9 的乙酰修饰,您可以在 UNIFI 科学库的修饰编辑器中定义“STG”的向前查找序列和/或“TAR”的向后查找序列。
- 通过示例向后查找序列,无论是与向前查找序列组合还是单独包含向后查找序列,修饰编辑器都会显示正确应用的修饰特异性。然而,在分析方法中使用时,它不会正确应用于在“分析方法”的“氨基酸修饰”部分中使用特定位点修饰的处理结果。
- 如果只包含一个向前查找序列,则修饰编辑器会显示正确应用的修饰特异性,并且它在方法的“氨基酸修饰”部分,在使用位点特异性修饰分析方法的处理结果中也能够正确应用。
环境
- waters_connect 3.2.0
- waters_connect 3.1.0
- 搭载 UNIFI 3.0.0 的 waters_connect
- 搭载 UNIFI 2.1.2 的 waters_connect
- 搭载 UNIFI 1.9.13 的 waters_connect
- UNIFI 1.9_SR4
原因
搜索词的定义不正确。
向前查找和向后查找功能需要在搜索定义中使用非常专属性的语法,否则无法正常工作。只输入氨基酸序列是不够的。遗憾的是,UNIFI 帮助文件 (UNIFI) 或 Administration 应用程序帮助文件 (waters_connect) 中均未对该语法进行解释
解决方法
向前查找
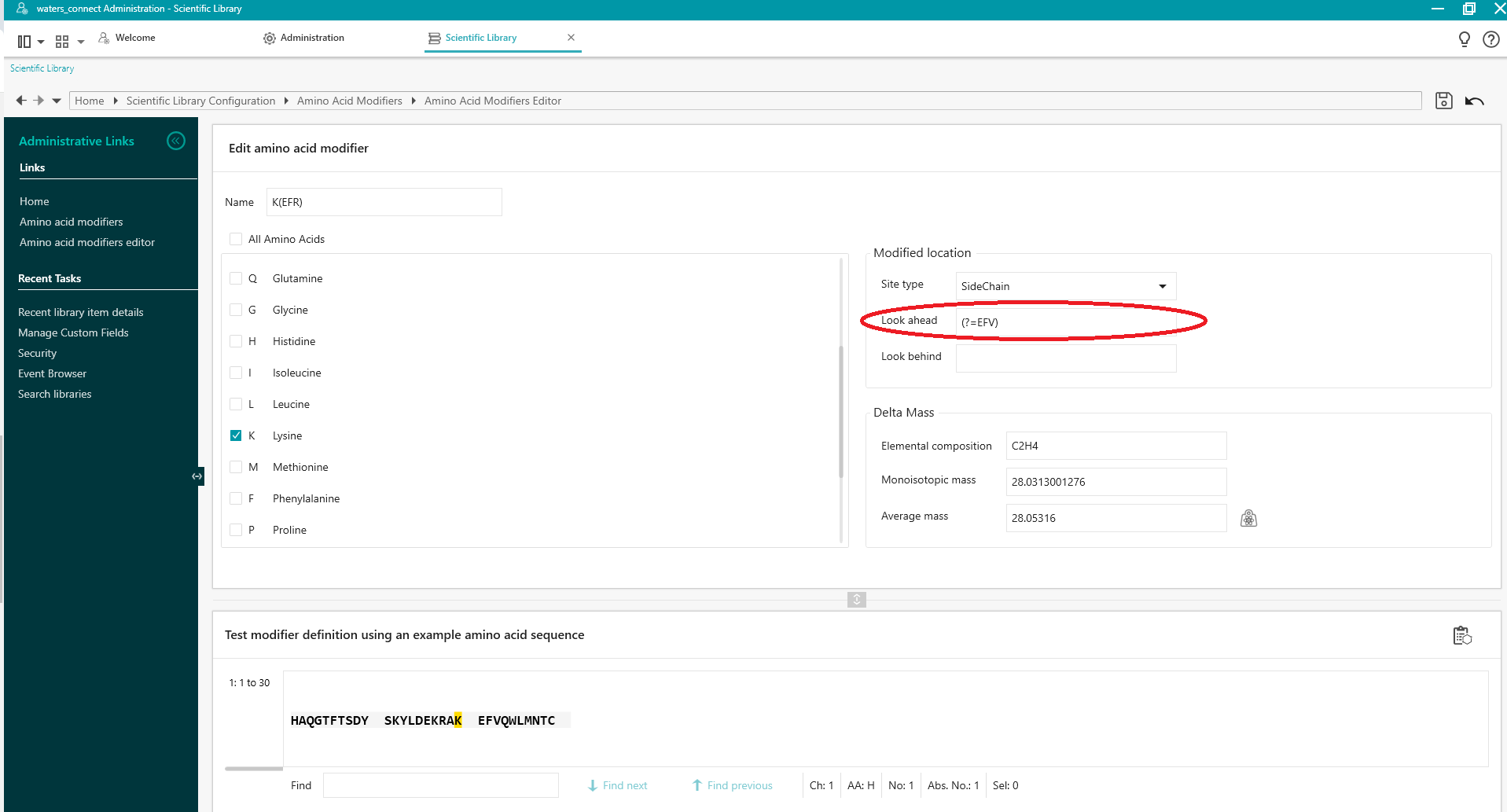
要在“应用到残基”之后使用氨基酸序列来定位该专属性残基的修饰,请使用以下语法:
(?=pattern),其中 pattern 为氨基酸的序列。
例如,要使用“向前查找”功能,仅将定义的修饰仅应用于序列 HAQGTFTSDYSKYLDEKRAKEFVQWLMNTC 中第 20 位的 K,请使用 (?=EFV) 搜索 K(?=EFV)(即,仅当 K 后跟 EFV 时,才将修饰应用于 K)。
向后查找
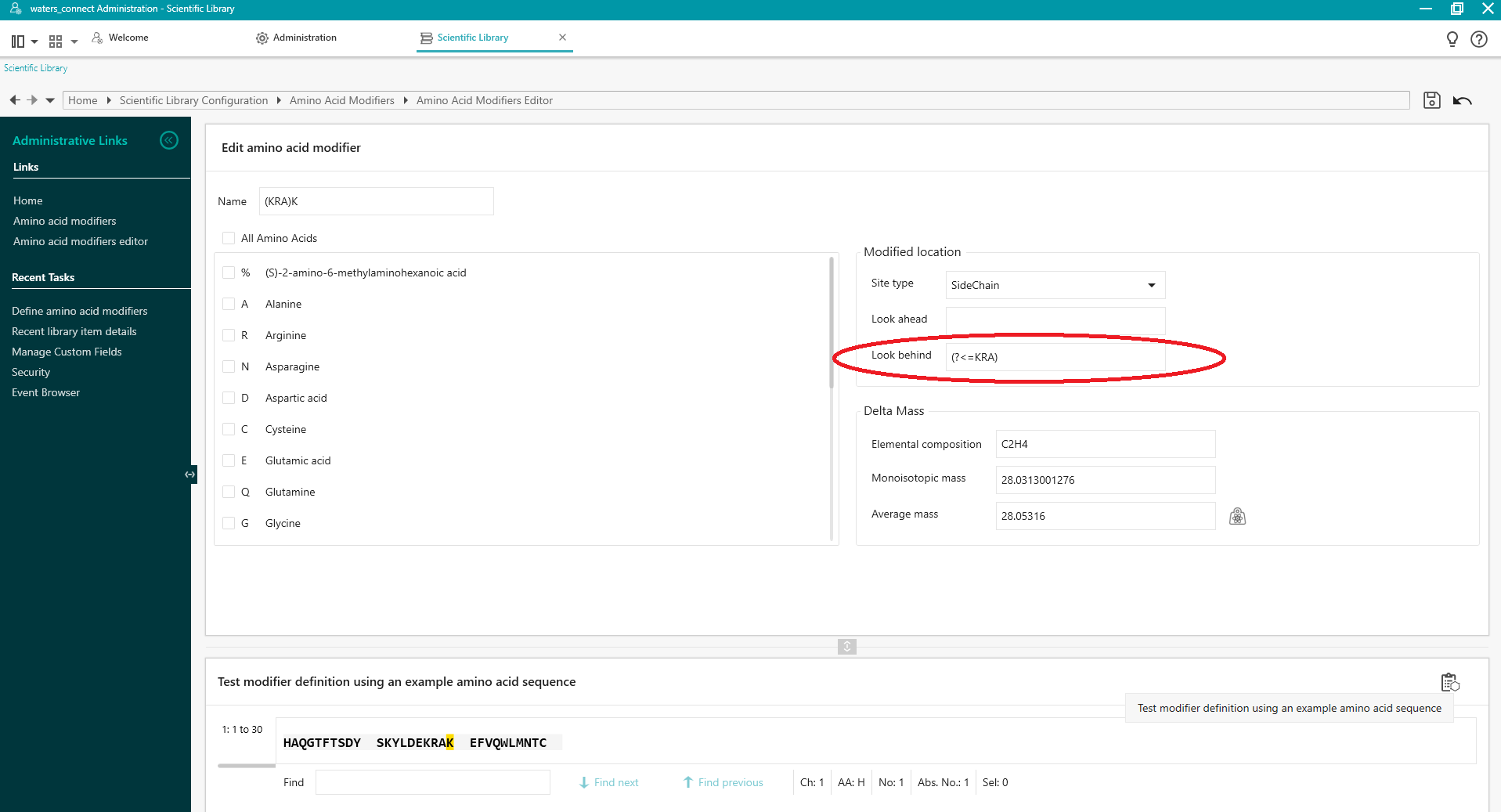
要在“应用到残基”之前使用氨基酸序列来定位该专属性残基的修饰,请使用以下语法:
(?<=pattern),其中 pattern 为氨基酸的序列。
例如,要使用“向后查找”将定义的修饰应用到序列 HAQGTFTSDYSKYLDEKRAKEFVQWLMNTC 中第 20 位的 K,请使用 (?<=KRA) 搜索 (?<=KRA)K(仅当前面带有 KRA 时,才将修饰应用于 K)。
附加信息
id118646, lysine, SUPUNIFI, UNIFISVR, UNIFISW18, UNIFOPT, UNIFQLIC, UNIFSW17, UNIFSW18, UNIFSW19, UNIFWGLIC, UNIFWKLIC