由于分辨率较差或峰形不佳,无法在 QTof(Xevo、Cyclic 或 Synapt)上进行校正 - WKB1813
文章编号: 1813点击此处访问英文版本文章
故障描述
- 在 Xevo 或 Synapt QTof 上进行校正或校正检查失败
- 校正能够运行,但某些峰不匹配,且不能手动添加(可能只有一个峰匹配)。
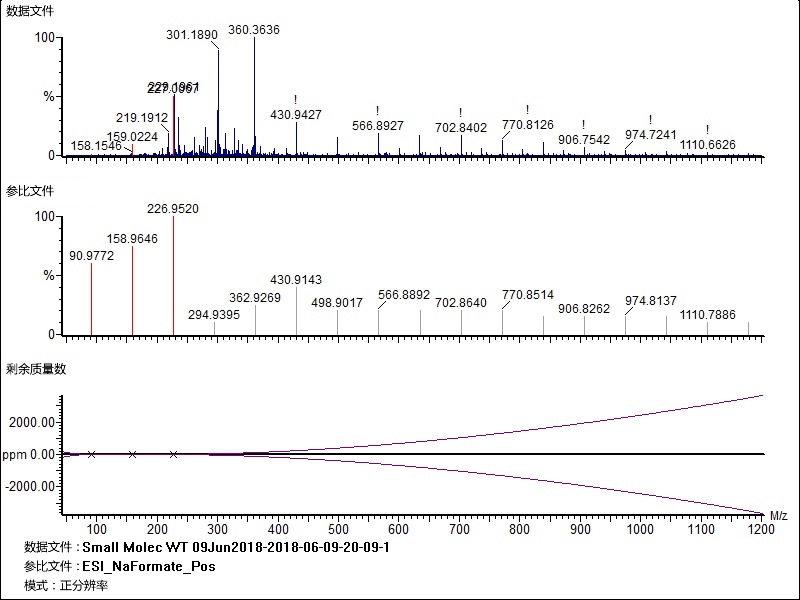
- 峰可能标记有感叹号 (!)
- 质量数较高时剩余质量数增加;请参见下图
- 质量数准确度差
- GluFib 上分辨率差
环境
- MassLynx 软件
- Intellistart
- UNIFI
- Synapt XS
- Synapt G2-Si
- Synapt G2-S
- Synapt G2
- Xevo G2-XS
- Xevo G2-S
- Xevo G2
- Xevo QTof
原因
谱图分辨率较差或峰形不佳
解决方法
- 打开 Tune 页面并选择 Setup > System View(设置 > 系统视图)调节分辨率。(密码为“access”。)
- 确保使用新制备的校正溶液或订购新样品。
- 在 Tune 页面上,选择 System > Acquisition Settings(系统 > 采集设置)。记下窗口左下方显示的 Resolution(分辨率)值,这是该分辨率模式的预期分辨率,然后关闭窗口。
- 通过放大锁定质量数峰并降低 LockSpray 毛细管电压或通过访问 System 2(系统 2)选项卡,在 pDRE Test(pDRE 测试)区域中选择 Attenuate(衰减)并调整 Transmission(传输),将计数调整为大约中低-e4 计数。如果您倾向于此操作,请右键单击 Tune 页面中的峰,然后选择 Intensity > Normalize Data(强度 > 归一化数据)。此设置使您能够在调节分辨率时查看强度是否增加或降低。
- 放大峰,以便轻松查看其形状。
- 在 System 2(系统 2)页面上,通过单击黑色数字右侧显示的滑块,然后使用向左箭头和向右箭头键(位于键盘上)来调节反射器网格电压 (reflectron grid)。调节获取最佳分辨率、峰形和灵敏度,分辨率优先级最高。
- 在 System 1(系统 1)页面上,通过单击黑色数字右侧显示的滑块,然后使用向左箭头和向右箭头键(位于键盘上)来调节推动电压偏置值 (pusher offset)。调节获取最佳分辨率、峰形和灵敏度,对称峰形优先级最高。
- 当分辨率接近或优于步骤 2 中记录的分辨率时,请打开控制台,然后选择 Normal mode(正常模式),路径为 Intellistart > Configure > Normal mode(Intellistart > 配置 > 正常模式),选择 Use calibration profile(使用校正配置文件),然后单击 Start(开始)。
- 浏览 uncal,然后单击 OK(确定)。
- 返回到 Tune 页面上的 System > Acquisition settings(系统 > 采集设置),单击 Calculate(计算),然后设置 Veff(有效电压)。指定所用参比的准确质量数(锁定质量数化合物,即阳离子模式中亮氨酸脑啡肽的 556.2771),并将 Tune 页面上观察到的质量数指定为保留两位小数的测量质量数。单击 Update(更新),等待几秒钟,然后关闭窗口。
- 如果之前执行了清除,请确保再次选中 Normalise(归一化),即右键单击 Tune 页面中的峰,然后选择 Intensity > Nomalise Data(强度 > 归一化数据)。您还可以将 LockSpray 毛细管电压重置为其正常值并取消选择 System 2(系统 2)选项卡上的 Attenuate(衰减)(如果适用)。
- 保存 Tune 设置和系统设置。
- 对要校正的所有其他离子模式重复此过程。
- 使用 Intellistart 校正经过调校的仪器,新的校正应能够提供更好的质量数准确度。
附加信息
制备新鲜的校正溶液
最好在校正之前调节分辨率,如果定期进行调节,则能够降低 Tune 工作量。
该分辨率可以通过 MS 控制台中的软件进行优化,通过从 LockSpray 流路注入亮氨酸脑啡肽,然后选择分辨率优化程序 -> 亮氨酸脑啡肽 MS LS 配置文件,再选择需要优化的模式。
id1813, capillary voltage, Capillary电压, enkephalin, Glu-血纤维蛋白肽, leucine, lockmass, MALDIG2SI, MLYNX, MLYNXV41, password, SUPMM, SYNAPTXS, SYNG2HD, SYNG2HDMAL, SYNG2MS, SYNG2MSMAL, SYNG2SHD, SYNG2SHDML, SYNG2SIHD, SYNG2SIMS, SYNG2SMS, SYNG2SMSML, XEVOG2QTOF, XEVOG2SQTF, XEVOG2STOF, XEVOG2TOF, XEVOG2XSTF, XEVOQTOF